2007/04/03 (火)
◆ [Software] URLを折り返すようにするFirefox拡張 [ARTIFACT@ハテナ系] - 21:02:56
なにーこんなんあったんか! Geckoエンジンのいつまでも直らない最大の欠点がこれで修正できるのか。あとはテーブル内の行間がしばしば狂う現象さえなんとかなればなぁ(NaTsuMi表組版参照。表示がおかしくならないときは何回か更新してみて下さい)。
◆ [Topics] 1GB DDR2メモリが6千円割れ - 17:19:02
そろそろ買い時か。でもこの辺のショップはPC2-6400だとまだ7千円台後半なんですよねぇ。まぁ私が年初に買ったときは1万1千円以上でしたから十分安いんですけど。あと、むしろ増やしたいのはGentoo入れて演算に使ってるマシンのPC-3200(DDR-400)だったり・・・。2GB積んでいても少しでかいデータセットで解析を走らせようとするとスワップしてしまう。
◆ [Life] ヲチ - 14:50:25
久しぶりに某スレをヲチ。すげぇ面白い。しかし学生を助けようとした(と思われる)某氏は災難ですな。
◆ [Science] Purvis (1995)の分岐年代推定法 - 14:20:50
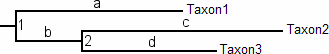
昨日書いたNatureのBininda-Emonds et al. (2007)で使われている分岐年代推定法の参考文献がPurvis (1995)だったんですが、JSTORだと見られなかったもののRoyal Societyのサイトからダウンロードできたのでさらっと眺めてみた。
Purvisが提案している方法(Local Molecular Clockと呼んでいるが、Local Molecular Clockに基づく実装は多種多様なので注意)は超単純。っつーか、岸野・長谷川著『分子系統学』のp156に書かれている方法とほとんど同じ。上の図で言えば分岐1が100万年前だとすると、分岐2の分岐年代は
100 * (((c + d) / 2) / ((a + (b + ((c + d) / 2))) / 2))
100 * (((c + d) / 2) / (b + ((c + d) / 2)))
となる(単位は万年)。ちなみに、Mean Path Length法(Bremer & Gustafsson, 1997; Britton et al., 2002)では
100 * (((c + d) / 2) / ((a + b + c + d) / 3))
100 * (((c + d) / 2) / ((a + (b + c) + (b + d)) / 3))
(ただし(c + d) / 2) < ((a + (b + c) + (b + d)) / 3))
である。
『分子系統学』を読んだときから思っていたんですが、この方法は分子進化速度の変化ははちゃめちゃになるのが欠点だと思う。系統樹を通して進化速度が一定に近い場合にはあまり問題にはならないでしょうけど、一定性が低い場合や突出した枝がある場合にはあまり良い方法ではないでしょう(MPLも同様)。
また、Purvisはもう一つ方法(Pure Birth Model)を提案している。以下のようなもの。
date of daughter branch = date of ancestor branch * (log(daughter clade size) / (log(ancestor clade size)))
だそうだ。上の図の場合で言えば分岐1が100万年前なら分岐2は
100 * (log2 / log3) = 63.1万年前(枝長に関係無く)
ってことか? こいつぁまたなんともTaxon Samplingによる影響をモロに受けそうな方法やな。哺乳類で全種網羅なら問題無いのかねぇ。まぁ哺乳類なら種の定義の曖昧さの影響は小さいのかもしれませんが、他の分類群には使えそうにないですね。
Bininda-Emonds et al. (2007)はLocal Molecular ClockとPure Birth Modelの両方を用いている。うーん、スンゲェ怪しい(笑)。
◆ [Tea] CHAMRAJ, FOP - 06:56:19

まぁまぁ。としか。悪くないんですがイマイチ印象に欠ける。ストレートよりもルピシアが薦めているようにレモンティーとかにした方がいいかも。
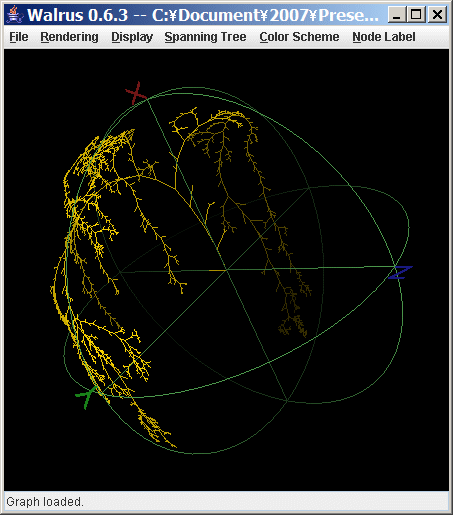
◆ [Science|Software] phylo3D - 06:26:45
NEXUSやNewick(PHYLIP)形式の樹形ファイルをWalrusで読み込み可能な形式に変換するソフト。Newick形式のファイルを変換するにはphylo3D.jarのあるディレクトリをカレントにして以下のようにコマンドを打つ(注:1行で入力する。UNIX系OSではセミコロンではなくコロンを使う)。
java -cp phylo3D.jar;ATVapp.jar;. phylo3D.tools.NHtoWalrus --inputFormat=NH input.nwk output.graph
これで生成されるoutput.graphファイルをWalrusで読み込んでレンダリングさせればよい。NEXUS形式ならinputFormatオプションでNEXUSと指定する。
ちなみに下の画像はランダムに生成したOTU1万のNewick形式系統樹をphylo3Dで変換してWalrusに描画させたもの。